서 론
1. 연구의 필요성
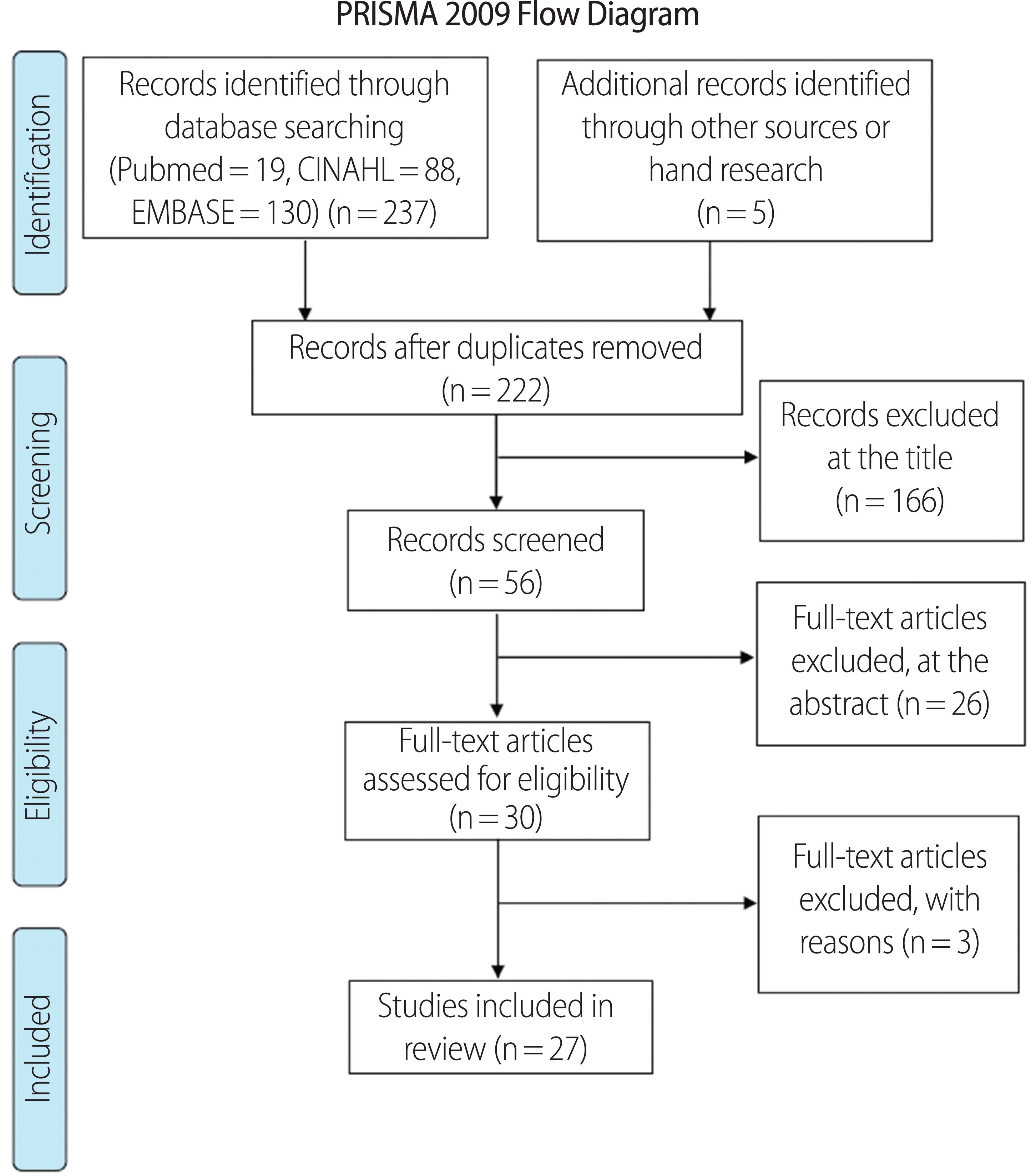
연구 방법
2. 연구 대상
3. 자료 수집
1) 문헌 검색
2) 자료 수집과 선별
연구 결과
1. 선택문헌의 일반적 특성
Table 1.
| Country | First author | Nation | Sample size | Age | Sex (M/F, no.) | Cancer type (no.) | CTx. regimen (no.) | Neurotoxicity measurement |
|---|---|---|---|---|---|---|---|---|
|
|
||||||||
| Western | Sereno (2017) | Spain | 94 | 66 (36-83) | 64/30 | CRC (69), biliary duct (4), pancreas (7), stomach (14) | XELOX (64), FOLFOX (17), others (13) | NCI-CTC, OSNS, TNSc |
| Argyriou (2013) |
Europe (3countries) |
200 | 63.7±8.9 | 120/80 | CRC (200), disease setting-adjuvant (122), -metastatic (78) | FOLFOX41) (102), FOLFOX62) (11), XELOX3) (87) | TNSc, NCI-CTC | |
| Cortejoso (2013) | Spain | 106 | 65 (median) | 62/44 | colon (72), rectum (34) grade of tumor-grade 1 (1), grade 2 (12), grade 3 (29), grade 4 (64) | FOLFOX (62), XELOX (40) | NCI-CTC | |
| Custodio (2013) | Spain | 387 | 61.73 (23-85) | 199/188 | colon (387), -stage II (108), stage III (279) | FOLFOX (209), XELOX (178) | NCI-CTC | |
| Cecchin (2012) | Italy | 144 | 59 (25-82) | 82/62 | colon (111), rectum (33), stage of disease -II (21), III (123) | FOLFOX4 (144) | NCI-CTC, OSNS | |
| Basso (2011) | Italy | 40 | 59 (median) | 25/15 | CRC (34), pancreatic ca. (5), bile ducts ca. (1) | FOLFOX4, FOLFOX6 | NCS | |
| Antonacopouplou (2010) | Greece | 55 | not reported | not reported | advanced CRC (55) | FOLFOX4 (55) | TNSc | |
| Mcleod (2010) | US | 520 | 61 (26-85) | 306/214 | m-CRC | IFL4) (114), FOLFOX (299), IROX5) (107) | NCI-CTC | |
| Zarate (2010) | Spain | 60 | 58 (37-75) | 45/15 | m-CRC | XELOX | NCI-CTC | |
| Boige (2010) | France | 346 | 68 (34-83) | 214/132 | m-CRC | FOLFOX | NCI-CTC | |
| Braun (2009) | UK | 280 (OXL) | 64 (27-85) | not reported | advanced CRC (280) | OxFU (OXL+fluorouracil) | NCI-CTC | |
| Argyriou (2009) | Greece | 62 | 62.4 (mean) | not reported | advanced CRC (62) | FOLFOX4 (62) | TNSc | |
| Paré (2008) | Spain | 126 | 66 (34-83) | 81/45 | m-CRC | OxFU (OXL+Fluorouracil) | OSNS | |
| Ruzzo (2007) | Italy | 166 | 66 (38-79) | 87/79 | CRC | FOLFOX4 | NCI-CTC, OSNS | |
| Gamelin (2007) | France | 135 | 62±11 | 93/42 | ||||
| Lecomte (2006) | France | 64 |
<63 (30) ≥63 (34) |
29/35 | CRC (58), pancreatic (4), gastric (2) | FOLFOX4 (46), FOLFOX76) (7), FOLFOX6 (6), GEMOX7) (4), TOMOX8) (1) | OSNS | |
| Stoehlmacher (2002) | US | 107 | 60 (24-83) | 77/30 | m-CRC | OxFU (OXL+Fluorouracil) | not reported | |
| Asian | Kanai (2016) | Japan | 465 | 64 (21-83) | 250/215 | colon | m-FOLFOX6 | NCI-CTC |
| Kumamoto (2013) | Japan | 63 | 65 (32-84) | 41/22 | colon/rectosigmoid (43), rectum (20) | m-FOLFOX6 | NCI-CTC | |
| Lee (2013) | Korea | 292 | 59 (30-76) | 183/109 | colon stage II (64), stage III (128) | FOLFOX | NCI-CTC | |
| Oguri (2013) | Japan | 70 | 65 (37-81) | 49/21 | CRC | FOLFOX6 | NCI-CTC | |
| Won (2012) | Korea | 343 | 56 (24-75) | 182/161 | colon (326), rectum (17), stage of disease: II (1), III (78), IV (264) | FOLFOX (264), XELOX (79) | NCI-CTC | |
| Hong (2011) | Korea | 52 | 63 (37-74) | 37/15 | m-CRC | XELOX | NCI-CTC | |
| Chen (2010) | China | 166 |
<50 (56) ≤50 (110) |
104/162 | colon (162), rectum (54) | FOLFOX | NCI-CTC | |
| Inada (2010) | Japan | 51 | 65 (37-81) | 34/17 | colon (22), rectum (27), other (2) | FOLFOX6 | NCI-CTC | |
| Kanai (2010) | Japan | 82 | 64 (41-80) | 51/31 | colon (47), rectum (35) | m-FOLFOX69) | OSNS | |
| Lai (2009) | China | 188 |
<50 (56) ≤50 (102) |
104/84 | colon (115), rectum (73) | FOLFOX4 | NCI-CTC | |
CTx=Chemotherapy; CRC=Colorectal cancer; XELOX=Oxaliplatin+capecitabine; FOLFOX=Oxaliplatin+leucovorin+5-FU; NCI-CTC=National Cancer Institute Common Toxicity Criteria; OSNS=Oxaliplatin-specific neurotoxicity scale; TNSc=Clinical version of the total neuropathy score; NCS=Nerve conduction study; m-CRC=Metastatic colorectal cancer; IFL=Irinotecan+5-FU+leucovorin; IROX=Oxaliplatin+irinotecan; GEMOX=Gemcitabine+oxaliplatin; TOMOX=Raltitrexed+oxaliplatin.
1)Oxaliplatin (85 mg/m2) administered as a 2-hour infusion on day 1; leucovorin (200 mg/m2) administered as a 2-hour infusion on day 1 and day 2; followed by a loading dose of 5-FU (400 mg/m2) IV bolus, then 5-FU (600 mg/m2) administered via ambulatory pump for a period of 22 hours on day 1 and day 2 every 2 weeks; 2)Oxaliplatin (85-100 mg/m2) administered as a 2-hour infusion on day 1; leucovorin (400 mg/m2) administered as a 2-hour infusion on day 1; followed by a loading dose of 5-FU (400 mg/m2) IV bolus on day 1, then 5-FU (2,400-3,000 mg/m2) administered via ambulatory pump for a period of 46 hours every 2 weeks; 3)Oral capecitabine (1,000 mg/m2) twice a day for 14 days plus oxaliplatin (130 mg/m2) on day 1 every 3 weeks; 4)Irinotecan 125 mg/m2 IV over 90 minutes; 5-FU 500 mg/m2 IV bolus; leucovorin 20 mg/m2 IV bolus weekly 4 weeks, every 6 weeks for 5 cycles; 5)Oxaliplatin 85 mg/m2 IV+irinotecan 200 mg/m2 IV, every 3 weeks; 6)Oxaliplatin 130 mg/m2 iv 2 hour day 1; lecovorin 400 mg/m2 iv 2 hour day 1; 5-fluorouracil 2,400 mg/m2 civ 46 hour, repeated every 2 weeks; 7) Gemcitabine 1,000 mg/m2 as a 10 mg/m2/min infusion on day 1, followed by oxaliplatin 100 mg/m2 as a 2-hour infusion on day 2, every 2 weeks; 8)Raltitrexed 3 mg/m2 plus oxaliplatin 100 mg/m2, both intravenously, on day 1 every 3 weeks; 9)DAY1- 85 mg/m2 oxaliplatin+400 mg/m2 bolus of 5-fluorouracil+200 mg/m2 L-leucovorin on day 1; 46 hours infusion of 2,400 mg/m2 5-fluorouracil every 2 weeks.
2. OXIPN의 발생률
Table 2.
| First author | Incidence/ severity of OXIPN | Candidate genes for OXIPN | Acute | Chronic | Incidence | Severity | Results |
|---|---|---|---|---|---|---|---|
|
|
|||||||
| Sereno (2017) | Moderate to severe (TNSc≥15): 48.9% Inexistent to mild (TNSc≤6): 51.1% | SCN9A-rs41268673, rs6746030, rs74401238 | N/A | N/A | ↓ | ↓ | S CN9A-rs6746030 was associated with protection for severe neuropathy (OR=0.39, 95% CI=0.16-0.96, p=.041) |
| Argyriou (2013) | Acute (after the discontinuation of Tx.): 84.5% (grade 1: 36.7%, grade 2: 27.2%, grade 3: 36.1%) | SCN4A-rs2302237 | (+) | (+) | ↑ |
↑ (acute) |
OR (95% CI; P) Incidence (G0 vs. G1-3) 2.39 (1.08-5.30; p=.028) Severity (G0-1 vs. G2-3) 2.04 (1.16-3.59; p=.013) |
| Chronic (at final f/u): 72.5% (grade 1: 34.5%, grade 2: 40.6%, grade 3: 24.8%) | SCN10A-rs12632942 | (+) | (-) | ↑ | (-) |
OR (95% CI; P) Incidence (G0 vs. G1-3) 0.45 (0.21-0.98; p=.045) Severity (G0-1 vs. G2-3) 0.84 (0.47-1.50; p=.560) |
|
| Cortejoso (2013) | Oxaliplatin based treatment peripheral neuropathy 12.3 % | ERCC1-rs11615 | N/A | N/A | (-) | (-) | D ata associated oxaliplatin-induced peripheral neuropathy was not reported in detail |
| Custodio (2013) | During treatment, severe OXIPN (grade 2-3) 31.0% |
CCNH-rs2230641 ABCG2-rs3114018 |
N/A | N/A | ↑ | ↑ |
OR (95% CI; p) Incidence CCNH-rs2230641 5.03 (1.06-2.41; p=.042) ABCG2-rs3114018 2.67 (0.95-4.41; p=.059) S everity CCNH-rs2230641 and/or ABCG2-rs3114018 2.46 (1.19-5.07; p=.014) |
| Cecchin (2012) | ncidence of neurotoxicity (within 48 hours before each cycle): 83.3% (any grade), 39% (grade≥2) | ABCC1-rs2074087, rs35587 | N/A | N/A | ↓ | N/A | G ≥2 toxicity/G0-1 minor allele frequency OR (95% CI) 0.43 (0.22-0.86) p=.170 (rs2074087) 0.47 (0.23-0.96) p=.038 (rs35587) |
| ABCC2-rs3740066, rs1885301, rs4148396, rs717620, ABCG2-rs2622604 | N/A | N/A | ↑ | N/A | G ≥2 toxicity/G0-1 minor allele frequency OR (95% CI) 3.06 (1.35-6.92) p=.007 (rs1885301) | ||
| Basso (2011) | Acute neurotoxicity (after every subsequent oxaliplatin administrations) | polymorphic CAG repeat motif within exon 1 of the SK3 gene | (+) | N/A | ↑ | N/A | C omparing G0-1/G2 Alleles (≤15/>15) OR 95% CI: 0.381 (0.247-0.590) p=.001 |
| G 0: 12 patients (30%); G1: 17 patients (43%); G2: 11 patients (27%) | |||||||
| Antonaco-pouplou (2010) | Chronic OXIPN (within a month after the completion of oxaliplatin based Tx.): 34/55 patients (61.8%) | ITGB3 (genotype C/C, T/T, C/T) | N/A | (+) | (-) | ↑ | I TGB3 at L33P genotype TT (incidence) No OXIPN: 47.6% OXIPN: 53% OR (95% CI) 2.43 (0.4-18.3) p=.490 |
| G1: 38.2%; G2: 61.8% | ITGB3 at L33P genotype TT (severity) G1: 30.8% G2: 66.7% OR (95% CI) 0.2 (0.1-1.2) p=.044 | ||||||
| McLeod (2010) | FOLFOX discontinuation rate 24% vs. 10%; 8/43 vs. 0/54 IROX patients (grade 3-4) | GSTP1 Ile105Val | (+) | N/A | ↑ | N/A | Patients with a GSTP1 I105V genotype of T/T were more likely to discontinue FOLFOX because of neurotoxicity (p=.010). In patients treated with IROX, only patients with T/T genotype experienced grade 3 neurotoxicity (p=.003) |
| Zarate (2010) | Neurotoxicity grade 1: 42%; grade 2: 25%; grade 3: 2% |
ERCC1 Asn118Asn GSTP1 Ile105Val GSTM1 deletion |
N/A | N/A | (-) | N/A | No specific result of neurotoxicity |
| Boige (2010) | Grade≥2 neurotoxicity arm A: 49%; arm B: 63% | ERCC1, ERCC2, GSTT1, GSTM1, GSTP1 | N/A | N/A | (-) | N/A | No statistically significant association was found between any genotype and FOLFOX-induced severe neurologic toxicity. |
| Braun (2009) | Not reported incidence of OXIPN seperated | ERCC2, GSTP1, MLH1 XRCC1 | N/A | N/A | (-) | (-) | Incidence of NCI-CTC G≥3 toxicity within 12 weeks of starting of oxaliplatin treatment regimen, by genotype TYMS-ER (p=.957), TYMS-1494 (p=.474) MTHFR (p=0.412) MLH1 (p=.327) UGT1A1 (p=.455) ABCB1 (p=.829) XRCC1 (p=0.423) GSTP1 (p=.301) ERCC2 (p=.915) |
| Argyriou A (2009) | Chronic OXIPN (following discontinuation of Tx.): 58.1%; grade 1: 38.9%; grade 2: 61.1% | SCN2A R19K | N/A | (-) | (-) | (-) |
Of patients without OXIPN (n=26), G/G: 80.8% (n=21) A/G: 19.2% (n=5) Of patients with OXIPN (n=36) G/G: 75% (n=27) A/G: 19.5% (n=7) A/A: 5.5% (n=2) G2 OXIPN (n=20) G/G: 72.7% (n=16) A/G: 11.1% (n=4) A/A: 9%(n=2) G1 OXIPN (n=14) G/G: 78.6% (n=11) A/G: 21.4% (n=3) |
| Paré (2008) | Neurotoxicity grade 1: 32.5%; grade 2: 52.4%; grade 3: 4% | GSTP1 Ile105Val | N/A | N/A | (-) | N/A | Grade 2 and 3 cumulative peripheral neuropathy was more common in patients with the Ile/Ile genotype than in patients with Ile/Val or Val/Val genotype, but the difference did not reach statistical significance (p=0.08). |
| Ruzzo (2007) |
(Evaluated after four cycles of treatment and then every 2 months) Grade 0: 43%; grade 1-2: 47%; grade 3-4: 10% |
GSTP1 exon 5 Val allele | (+) | (+) | N/A | ↑ | The frequencies of GSTP1-105A/A, A/G, and G/G genotypes: 63%, 34%, 2% (78 patients with G1-2 neurotoxicity) and 23%, 30%, 47% (17 patients with G3 neurotoxicity) (p<.001). |
| Gamelin (2007) | Acute neuropathy (n) Grade 0: 50; grade 1: 58; grade 2: 20; grade 3: 7 |
AGXT Ile340Met AGXT pro11Leu GSTP1 Ile105Val |
(+) | (+) | (+) | (+) |
Significant association (p<.001) with CIPN severity for AGXT Pro11Leu C/T and T/T vs. C/C, and for AGXT Ile340Met A/G and G/G vs. A/A |
| Chronic neuropathy (n) Grade 0: 105; grade 1: 2; grade 2: 15; grade 3: 13 | |||||||
| Lecomte (2006) |
Cumulative peripheral neuropathy Grade 1: 42%, grade 2: 27%, grade 3: 23% |
GSTP1 exon 5 Val allele | N/A | (+) | ↓ | N/A | Patients with G3 OXIPN (lle/lle; 87%, lle/Val;13%, Val/Val; 0, lle/Val+Val/Val;13%) Patients without G3 OXIPN (lle/lle; 53%, lle/Val;37%, Val/Val; 10%, lle/Val+Val/Val;47%)→OR 95% CI; 5.75 (1.08-30.74), p=.020 |
| Stoehlmacher (2002) | 10 patients ith grade 3-4 neurological toxicity (not indicated lower grades) |
G STP1 Ile105Val, GSTM1 deletion, GSTT1 deletion |
N/A | N/A | (-) | N/A | No specific result of neurotoxicity |
| Kanai (2016) |
Not reported the time point of assessment Grade 1: 61.9%, grade 2: 31.6%, grade 3: 6.5% |
A BCG2, ACYP2, BTG4, CCNH, CAMK2N1, DLEU7, FARS2, FOXC1, GSTP1, ITGA1, TAC1, XRCC1 | N/A | N/A | (-) | (-) | Comparison of grade 0/1 with grade 2/3 or grade 3 showed no significant association with any of the 12 SNP markers after adjustment for total dose of oxaliplatin. |
| Kumamoto (2013) | Grade 2: 66.7%; grade 3: 3.2% |
GSTP1-105 A/G, G/G GSTM1-negative |
(+) | N/A | ↑ | ↑ | A statistically significant correlation between the icidence of peripheral neuropathy higher than grade 2 and the GSTP1-105 (p=.030) and GSTM1 (p=.020) genotypes was determined. |
| Lee (2013) |
Sensory neuropathy (assessed every 2 weeks during treatment) (257/292 patients, 88%) Grade 1: 69.1%; grade 2: 15.8%; grade 3: 1.7%; grade 4: 0% |
XRCC1 | (+) | N/A | ↓ | (-) |
Patients harboring the XRCC1 23885GG experienced less G2-4 (12.9% vs. 21.7%, p=.050, /OR=0.52 95% CI 0.27-0.99, p=.046). Time to the onset of G2-4 neuropathy was later in patients haboring XRCC1 23885GG genotypes (HR 0.56, 95% CI 0.32-0.99, p=.041) compared to AA/AG |
| Oguri (2013) |
Acute neurotoxicity (n=65) (within a week after infusion of OXL) Grade 1: 71%, grade 2: 17%, grade 3: 4% Chronic neurotoxicity (n=48) Grade 1: 37%, grade 2: 21%, grade 3: 10% |
TAC1 (rs10486003) FARS2 (rs17140129) ERCC1 (rs11615) |
N/A | (+) | ↑ | ↑ |
FARS2, risk of severe chronic neuropathy compared to A/A, A/G & GG genotype (p=.034, OR=6.5 CI 95% 1.2-35.7). TAC1&ERCC1, median number of Tx. Cycles at onset- C/C: C/T and T/T=6:8 (p=.049) |
| Won (2012) |
Severe OXIPN (evaluated on day 1 of each chemotherapy cycle) 39/96 patients (41%) 85/247 patients (34%): the discovery set and the validation set, respectively. |
TAC1, FOXC1, GMDS, ITGA1, PELO, ACYP2, TSPYL6, DLEU7 | (+) | N/A | ↑or↓ | N/A |
Genes OR (95% CI) TAC1 0.32 (0.19-0.52) FOXC1, GMDS 2.27 (1.58-3.26) ITGA1, PELO 0.32 (0.19-0.54) ACYP2, TSPYL6 2.43 (1.61-3.68) DLEU7 0.50 (0.35-0.72) |
| Hong (2011) |
Sensory neuropathy Grade 1: 57.7%; grade 2: 7.7%; grade 3-4: 7.7% |
GSTP1-Ile105Val | (+) | N/A | ↑ | N/A | GSTP1-Ile105Val was associated with neurotoxicity. Patients with A/G or G/G genotypes experienced more frequent G2/3 sensory neuropathy (35.7% in A/G or G/G vs. 8.1% in A/A, p=.030). |
| Chen (2010) |
lle/lle vs. lle/Val or Val/Val After 4cycles: grade 0-2 99.2%/97.6% grade 3-4 0.8%/2.4%; After 8cycles: grade 0-2 92.8%/80.5% grade 3-4 7.2%/19.5%; After 12 cycles: grade 0-2 85.6%/63.4% grade 3-4 14.4%/36.6% |
GSTP1 Val105 | (+) | N/A | ↑ | ↑ |
Ile/Ile vs. Ile/Val or Val/Val After 8 cycles- G3-4 Val105 allele variants (7.2% vs. 19.5%, p=.020). After 12 cycles- (14.4% vs. 36.6%, p<.010). |
| Inada (2010) |
Acute neuropathy (n=49) Grade 1: 38 patients (75%) grade 2: 8 (16%) grade 3: 3 (6%). Chronic neuropathy (n=36) G rade 1: 21 patients (41%) grade 2: 8 (16%) grade 3: 7 (14%) |
ERCC1, GSTP1 | N/A | (+) | (+) | (-) |
ERCC1 C118T median number of Tx. Cycles at onset C/T&T/T vs. C/C=6:7 (p=.016) G STP1 Ile105Val median number of Tx. Cycles at onset Ile/Ile vs. Ile/Val&Val/Val 6:9 (p=.032) |
| Kanai (2010) | 44 patients (54%) developed grade 2/3 oxaliplatin induced neuropathy during the 2 weeks after completion of the 6th cycle of m-FOLFOX6 | GSTP1, AGXT | (-) | N/A | (-) | (-) |
GSTP1 Ile105Val (G1/G2/G3, %) Ile/Ile (52/47/0) Ile/Val (32/64/4) Val/Val (50/50/0) p=.160 AGXT Ile340Met (G1/G2/G3, %) Ile/Ile (48/51/1) Ile/Met (45/55/0) Met/Met (0/100/0) p=1.000 |
| Lai (2009) | Presence of grade 3-4 oxaliplatin neuropathy (after 2, 4, 6 courses of Treatment) 30/188 (16%) | XPD K751Q | N/A | N/A | (-) | N/A |
Incidence Lys/Lys vs. Lys/Gln 13.3% vs. 16.5%, (p=.670) |
OXIPN=Oxaliplatin induced peripheral neuropathy; TNSc=Clinical version of the total neuropathy score; SCN=Sodium voltage-gated channel; N/A=Not applicable; OR=Odds ratio; CI=Confidence interval; Tx=Treatment; f/u=Follow-up; ERCC=Excision repair cross-complementation group; CCNH=Cycline H; ABCG=ATP-binding cassette subfamily; ABCC=ATP-binding cassette subfamily; CAG=Cytosine-adenineguanine; SK=Small conductance calcium-activated potassium channel; ITGB=Integrin beta; FOLFOX=Oxaliplatin+leucovorin+5-FU; IROX=Oxaliplatin+ irinotecan; GSTP=Glutathione-S transferases pi; GSTM=Glutathione-S transferases Mu; GSTT=Glutathione S-transferases theta; MLH=NutL homolog; XRCC=X-ray repair cross-complementing protein; NCI-CTC=National Cancer Institute Common Toxicity Criteria; TYMS-ER= Thymidylate synthase-enhancer region; TYMS=Thymidylate synthase; MTHFR=Methylene tetrahydrofolate reductase; UGT1A1=Uridine diphosphate glycosyltransferases 1A1; ABCB= ATP-binding cassette sub-family B; AGXT=Alanine-glyoxylate aminotransferase; ACYP=Acylphosphatase; BTG=B-cell translocation gene; CAMK2N1=Calcium/calmodulin-dependent protein kinase II inhibitor 1; DLEU=Deleted in lymphocytic leukemia; SNP=Single nucleotide polymorphisms; FARS=Phenylalanyl-tRNA synthetase; FOXC1=Foxhead box C1; ITGA=Integrin alpha; TAC=Tachykinin precursor; HR=Hazard ratio; GMDS=GDPmannose 4,6-dehydratase; PELO=Pelota homolog; TSPYL=Testis-specific Y-encoded-like protein; XPD=Xeroderma pigmentosum D ortholog.



 PDF Links
PDF Links PubReader
PubReader Full text via DOI
Full text via DOI Download Citation
Download Citation Print
Print